As escolhas de uma pessoa durante a vida podem alterar seu DNA, literalmente.
As alterações genéticas que ocorrem nas células humanas ao longo da vida de uma pessoa, conhecidas como mutações somáticas, são o que causam a grande maioria dos casos de câncer. Elas podem ser provocadas por fatores ambientais ou comportamentais, como exposição a raios ultravioleta ou radiação, consumo de álcool ou tabagismo.
Com o uso das GPUs NVIDIA na análise da assinatura, ou impressão digital molecular, dessas mutações, os pesquisadores podem entender melhor as causas conhecidas do câncer, descobrir novos fatores de risco e identificar os motivos pelos quais determinados tipos de câncer são mais comuns em certas regiões do mundo do que em outras.
A equipe do projeto Mutographs, um grupo de pesquisa internacional financiado pela Cancer Research UK, da iniciativa Cancer Grand Challenges está usando modelos de machine learning acelerados por GPUs NVIDIA para estudar o DNA dos tumores de 5 mil pacientes com cinco tipos de câncer: pâncreas, rim, colorretal e dois tipos de câncer de esôfago.
Com os potentes sistemas NVIDIA DGX, pesquisadores do Instituto Wellcome Sanger, líder mundial em genômica, e da Universidade da Califórnia, em San Diego, colaboraram com desenvolvedores da NVIDIA para garantir uma aceleração de mais de 30 vezes na execução do software de machine learning SigProfiler.
“Projetos de pesquisa como o Mutographs do Grand Challenge são exatamente isso: grandes desafios que ampliam os limites de nossa capacidade”, afirmou Pete Clapham, Líder do Grupo de Apoio de Informática do Instituto Wellcome Sanger. “Os sistemas NVIDIA DGX oferecem uma aceleração considerável que garante que a equipe do Mutographs não só atenda às demandas computacionais do projeto, mas também o desenvolva ainda mais, gerando, com eficiência, resultados que antes eram impossíveis.”
Investigação Molecular
Assim como as pessoas têm uma impressão digital única, as mutações somáticas causadoras de câncer têm padrões únicos que aparecem no DNA de uma célula.
“Na cena de um crime, os investigadores coletam as impressões digitais e comparam-nas com as de um banco de dados para encontrar uma correspondência”, explicou Ludmil Alexandrov, Líder Computacional do projeto e Professor Assistente de medicina celular e molecular da UCSD. “Da mesma forma, podemos analisar a impressão digital molecular das células coletadas na biópsia de um paciente e ver se ela está relacionada a fatores de risco, como tabagismo e exposição a raios ultravioleta.”
As fontes de algumas mutações somáticas são conhecidas, como as mencionadas por Alexandrov. Porém, o modelo de machine learning consegue identificar outros padrões de mutação que ocorrem repetidamente em pacientes com um tipo específico de câncer, mas sem fonte conhecida.
Quando isso acontece, Alexandrov se reúne com outros cientistas para testar as hipóteses e realizar experimentos em grande escala para descobrir o agente causador de câncer.
A descoberta de um novo fator de risco pode ajudar a melhorar a prevenção do câncer. Em 2018, pesquisadores identificaram a assinatura mutacional de um tipo de câncer de pele relacionada ao uso de um imunossupressor, condição que agora consta na bula como um dos possíveis efeitos colaterais, ajudando médicos a monitorar melhor os pacientes que estão sendo tratados com o medicamento.
Realização de Análises Rápidas de Dados Globais
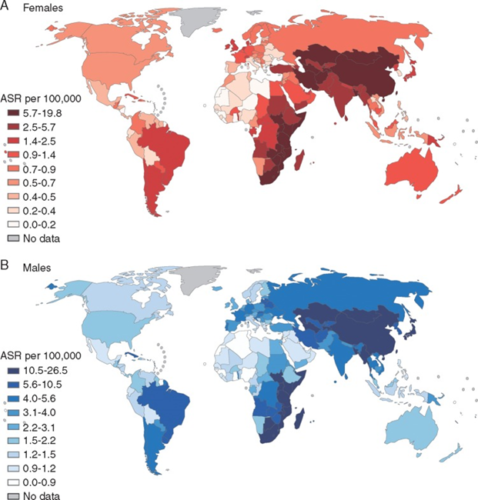
Nos casos em que a fonte da assinatura mutacional é conhecida, os pesquisadores podem analisar tendências na ocorrência de tipos específicos de mutações somáticas (e seus tipos de câncer correspondentes) em diferentes regiões do mundo e ao longo do tempo.
“Alguns tipos de câncer são muito comuns em uma região do mundo e muito raros em outras. Além disso, quando as pessoas migram de um país para outro, elas passam a correr o risco de câncer do país para o qual migraram”, revelou Alexandrov. “Essa informação nos mostra que os fatores são principalmente ambientais.”
Os pesquisadores do projeto Mutographs estão estudando uma mutação somática relacionada ao câncer de esôfago, uma condição que alguns estudos associaram ao consumo de bebidas muito quentes, como chá ou mate.
O câncer de esôfago é muito mais comum no leste da América do Sul, na África Oriental e na Ásia Central do que na América do Norte ou na África Ocidental. A identificação do fator ambiental ou de estilo de vida que aumenta o risco de surgimento de câncer nas pessoas pode ajudar na prevenção e na detecção precoce de casos futuros.
Os pesquisadores do Mutographs colaboraram com a NVIDIA para acelerar as partes mais demoradas do framework de AI SigProfiler em GPUs NVIDIA. Ao processar tarefas do pipeline com precisão dupla em sistemas NVIDIA DGX, a equipe observou uma aceleração de mais de 30 vezes em relação ao uso da CPU do hardware. Com a precisão única, segundo Alexandrov, o SigProfiler funciona muito mais rapidamente, acelerando as tarefas em até 50 vezes.
Com o software otimizado do sistema DGX e a tecnologia de interconexão NVLink, também é possível dimensionar modelos de AI em todas as oito GPUs NVIDIA V100 Tensor Core do sistema, garantindo o desempenho máximo no desenvolvimento e na implantação de modelos.
Em um estudo publicado neste ano na revista Nature, a equipe de Alexandrov analisou dados de mais de 20 mil pacientes de câncer, uma tarefa que levava quase um mês.
“Agora, com o NVIDIA DGX, podemos realizar essa mesma análise em menos de um dia”, afirmou. “Assim, poderemos realizar muito mais testes, validações e explorações.”
Inscreva-se no boletim informativo da NVIDIA sobre a área da saúde.
Crédito da imagem principal: Instituto Wellcome Sanger