Cientistas e pesquisadores da área da saúde, que antes eram limitados pelo número de amostras que podiam estudar em um laboratório experimental ou pela qualidade dos microscópios para observar as células, estão usando ferramentas computacionais potentes para extrair informações de um grupo cada vez maior de dados biológicos.
Para apoiar essa revolução da biologia digital, são usados sistemas computacionais de alto desempenho e frameworks de software de domínios específicos.
Dois supercomputadores da lista TOP500 dos sistemas mais potentes recentemente anunciados, o Cambridge-1, que é da NVIDIA e é focado na área da saúde, e o BioHive-1, da empresa de biotecnologia Recursion, são baseados na arquitetura de referência NVIDIA DGX SuperPOD.
Além disso, instituições de pesquisa médica, empresas farmacêuticas e startups de biotecnologia de todo o mundo usam o NVIDIA Clara Parabricks, um conjunto de bibliotecas e aplicações de referência de genômica, para aprimorar workflows de sequenciamento de última geração.
Em junho passado, a Mingma Biotechnology, com sede em Xangai, se tornou a empresa com o primeiro laboratório de pesquisa da China a usar o Clara Parabricks Pipelines no projeto de medicina de precisão da organização. Ainda neste ano, foram lançadas iniciativas de genômica de grande escala na Tailândia e no Japão. Além disso, recentemente, a startup de terapia genética Greffex adotou o Parabricks Pipelines para acelerar seu projeto de desenvolvimento de uma vacina universal contra a gripe.
Identificando Insights Genômicos para Estudos Populacionais
O Parabricks Pipelines acelera projetos baseados em DNA e RNA em até 50 vezes nas GPUs NVIDIA, permitindo que os cientistas extraiam o máximo de informações úteis possível das centenas de terabytes de dados de instrumentos gerados diariamente. Essa aceleração é vantajosa principalmente para instituições de saúde pública e laboratórios de pesquisa que realizam estudos populacionais com dezenas de milhares de genomas que precisam ser analisados.
A Mingma Biotechnology adotou o Parabricks Pipelines e GPUs NVIDIA T4 Tensor Core para acelerar seu trabalho na análise de dados de sequenciamento e multiômica. A empresa oferece a instituições médicas, empresas farmacêuticas e pesquisadores informações genômicas para a pesquisa de doenças e o desenvolvimento de medicamentos.
No National Biobank of Thailand, um sistema NVIDIA DGX A100 é usado na Genomics Thailand, uma iniciativa para estabelecer a medicina genômica como um serviço de saúde comum no país. A instituição de pesquisa está usando o Parabricks Pipelines para analisar variações genéticas de 50 mil dados do sequenciamento do genoma completo de voluntários tailandeses.
A combinação do sistema DGX com o Parabricks Pipelines reduziu em quatro meses o tempo de processamento de dados do genoma completo do projeto. As informações desse trabalho ajudarão os pesquisadores a analisar melhor a variação genética específica da população tailandesa.
No Japão, o Human Genome Center da Universidade de Tóquio lançou recentemente o SHIROKANE, o supercomputador mais rápido do país para ciências biomédicas. O sistema DGX A100 executa o Parabricks Pipelines para sequenciar o genoma completo de 92 mil pacientes, criando um banco de dados que é fundamental para os projetos de medicina de precisão para câncer e doenças sem cura.
Possibilitando o Sequenciamento Clínico e a Descoberta de Medicamentos
É possível configurar o conjunto de ferramentas genéticas Parabricks Pipelines para atender às necessidades específicas de cada laboratório. Os pesquisadores executam cargas de trabalho do Parabricks Pipelines em sistemas com GPUs NVIDIA que vão desde workstations até clouds acelerados por GPU e alguns dos supercomputadores mais rápidos do mundo.
Poucas semanas depois de começar a usar uma workstation de ciência de dados com GPUs NVIDIA RTX, a Greffex, com sede em Houston, adotou o Parabricks Pipelines e o NVIDIA Clara Discovery para promover seus projetos de desenvolvimento de uma vacina universal contra a gripe.
A startup usa uma combinação de sequenciamento genômico, ferramentas de dinâmica molecular e pesquisas em laboratórios experimentais para estudar como as cepas do vírus influenza evoluem com o tempo e como essas mutações afetam a eficácia da vacina.
Para monitorar as mudanças nos vírus, a Greffex coleta dezenas de milhares de genomas gripais de todo o mundo e realiza grandes alinhamentos de sequências nas GPUs NVIDIA RTX 8000 para identificar onde o código genético do organismo está mudando. Com a execução de cargas de trabalho genômicas em GPUs, a empresa economiza até 13 horas por amostra, além de permitir que a equipe teste amostras novamente com diferentes parâmetros para ajustar os resultados de alinhamento.
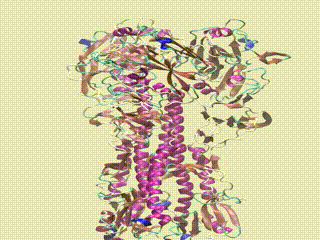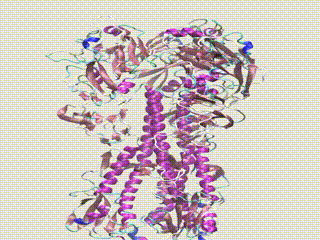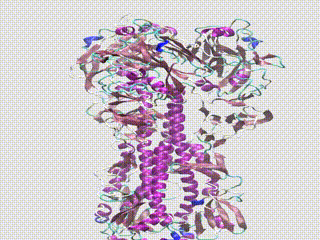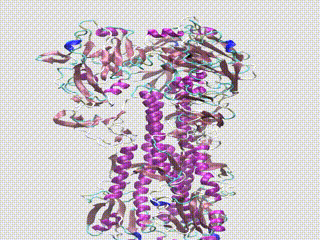
Depois de identificadas as variantes genéticas, os cientistas da Greffex usam a dinâmica molecular para visualizar como as alterações genéticas mudam a forma física do vírus da gripe. Eles procuram mutações divergentes, que fazem com que o vírus ganhe uma forma que não se liga tão bem aos anticorpos criados a partir da vacina.
“O processo de otimização da estrutura proteica para uma vacina que se liga não apenas à cepa atual da gripe, mas a diversas outras é muito longo e caro. Com métodos computacionais, podemos ter uma noção do que provavelmente funcionará antes de testar em laboratórios reais. É como usar um bisturi em vez de um martelo”, afirmou Daniel Preston, Cientista de Bioinformática da Greffex.
Sobre o NVIDIA Clara Parabricks
O NVIDIA Clara Parabricks oferece acelerações por GPU para o Genome Analysis Toolkit padrão do Broad Institute, além de ferramentas comuns como o mecanismo genético DeepVariant da Google. Em execução em GPUs NVIDIA A100 Tensor Core, o Parabricks diminui para 23 minutos o tempo da análise secundária de um genoma humano completo para a previsão de variantes germinativas do DNA, o que em um sistema com CPU leva mais de 20 horas.
A partir de leituras do sequenciamento do DNA, os Pipelines do Clara Parabricks podem alinhar, classificar, filtrar e prever variantes para a detecção de variantes somáticas e germinativas, além de oferecer suporte a aplicações baseadas no RNA. As variantes germinativas são herdadas da ancestralidade de uma pessoa, e as mutações somáticas ocorrem em células humanas ao longo da vida de um indivíduo e podem causar câncer.
A versão 3.6 dos Pipelines do Parabricks , lançada no próximo mês, oferecerá mais ferramentas para a previsão de variantes somáticas, o que disponibiliza aos pesquisadores informações sobre a oncologia de precisão, e de outras previsões de variantes germinativas, úteis para pesquisas sobre doenças complexas como o autismo.
O pipeline de novas variantes germinativas foi desenvolvido em colaboração com pesquisadores da School of Medicine da Universidade de Washington. A técnica reduz para menos de uma hora o tempo de execução para analisar os dados do genoma e identificar novas variantes em um tipo de família ou trio de pais e filho(a).
Comece a usar os Pipelines do NVIDIA Clara Parabricks para realizar análises aceleradas do genoma no NGC ou no AWS Marketplace.
A imagem principal mostra um alinhamento estrutural de 17 proteínas H1 hemaglutinina ao longo de 102 anos com anticorpos vinculados simulados. Os segmentos coloridos correspondem a diferentes tipos de mutação nas proteínas. A imagem foi cedida pela Greffex usando modelos do RCSB Protein Data Bank.