Descobrir medicamentos é como encontrar a peça certa de um quebra-cabeça, só que com 1060 do tamanho de uma molécula. As ferramentas de AI ajudam os pesquisadores a limitar as opções, selecionando um subconjunto de peças com o formato e a cor certos que serão testadas.
Um fármaco eficaz se liga à enzima ou ao receptor desejado, um ponto específico na célula com a qual ele está tentando interagir. Como a peça ideal de um quebra-cabeça, quando um medicamento é eficiente, ele se encaixa perfeitamente, apresentando a forma, a flexibilidade e a energia de interação (a distribuição de carga elétrica de uma molécula) certas para se ligar ao elemento.
Algumas drogas são agonistas, como a morfina, que ativa os receptores desejados do cérebro para produzir um efeito analgésico. Outras, como a insulina, são antagonistas e, em vez disso, inibem ou bloqueiam os receptores.
Pesquisadores de todo o mundo estão na corrida para encontrar um candidato a medicamento eficaz para evitar a infecção e a replicação do SARS-CoV-2, o vírus causador da COVID-19. Com GPUs NVIDIA, eles estão acelerando o processo de descoberta de medicamentos, seja para o desenho de medicamentos baseado na estrutura, o acoplamento molecular, modelos generativos de AI, a triagem virtual ou a triagem de grande escala.
Identificando as Proteínas Desejadas com a Genômica
Os pesquisadores precisam saber por onde começar para desenvolver um fármaco eficaz. Na via de uma doença, uma cadeia de sinais entre moléculas que ativam diferentes funções celulares, podem existir milhares de proteínas interagentes. As análises genômicas podem gerar informações valiosas para os pesquisadores, ajudando-os a identificar as proteínas promissoras que devem ser priorizadas com um composto de medicamento.
Com o kit de ferramentas de análise genômica NVIDIA Clara Parabricks, os pesquisadores podem sequenciar e analisar genomas até 50 vezes mais rapidamente. Como o vírus causador da pandemia de COVID-19 se espalha com facilidade, garantir resultados em horas, e não em dias, pode ter um impacto extraordinário na compreensão do vírus e no desenvolvimento de tratamentos.
Até hoje, centenas de instituições, como hospitais, universidades e centros de supercomputação, de 88 países baixaram o software para acelerar seus projetos, tanto para sequenciar o genoma viral quanto para sequenciar o DNA de pacientes com COVID-19 e descobrir por que alguns são mais afetados pelo vírus do que outros.
Outro método, a criomicroscopia eletrônica, usa microscópios eletrônicos para observar proteínas diretamente, podendo usar GPUs para reduzir o tempo de processamento de conjuntos de dados grandes e complexos.
Com o CryoSPARC, um software acelerado por GPU desenvolvido pela startup de Toronto Structura Biotechnology, pesquisadores dos Institutos Nacionais de Saúde e da Universidade do Texas em Austin criaram o primeiro mapa 3D de escala atômica do coronavírus, trazendo uma visão detalhada das proteínas da espícula do vírus, um alvo importante para vacinas, anticorpos e diagnósticos.
Triagem de Compostos Acelerada por GPU
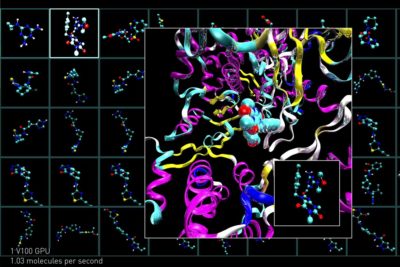 Depois de identificar a proteína desejada, os pesquisadores procuram compostos de medicamentos com as propriedades ideais para se ligarem a ela. Para saber a eficácia dos candidatos a medicamentos, os pesquisadores podem realizar a triagem deles virtualmente e em laboratórios físicos.
Depois de identificar a proteína desejada, os pesquisadores procuram compostos de medicamentos com as propriedades ideais para se ligarem a ela. Para saber a eficácia dos candidatos a medicamentos, os pesquisadores podem realizar a triagem deles virtualmente e em laboratórios físicos.
A startup de descoberta de medicamentos Atomwise, participante do programa NVIDIA Inception desenvolveu a AtomNet, uma rede neural convolucional que consegue realizar a triagem de mais de 16 bilhões de compostos em menos de dois dias em GPUs NVIDIA hospedadas no cloud. A triagem virtual pode acelerar o tempo necessário para descobrir candidatos a medicamentos promissores para testes adicionais, um processo que poderia levar meses ou anos e custaria milhões de dólares.
Sediada em Nova York, a Schrödinger cria softwares de descoberta de medicamentos que conseguem modelar as propriedades de possíveis fármacos. Usada pelas maiores empresas biofarmacêuticas do mundo, a plataforma Schrödinger oferece aos usuários a capacidade de sintetizar e determinar a afinidade de ligação de um fármaco em GPUs NVIDIA Tensor Core em menos de uma hora e com apenas alguns dólares de custo de computação em vez de milhares de dólares usando métodos tradicionais.
Modelos Generativos de AI para a Descoberta de Medicamentos
Em vez de avaliar um conjunto de dados de candidatos a medicamentos conhecidos, o modelo generativo de AI começa do zero. A startup Elix , Inc., sediada em Tóquio e participante do programa de aceleração virtual NVIDIA Inception usa modelos generativos treinados em sistemas NVIDIA DGX Station para identificar estruturas moleculares promissoras. Algumas moléculas propostas pela AI não se encontram na natureza, então é preciso usar outras redes neurais para determinar a viabilidade da sintetização desses candidatos no laboratório.
Com a DGX Station, a Elix consegue acelerar o treinamento de modelos generativos em até 6 vezes, o que levaria uma semana ou mais para chegar a um resultado estável ou para garantir a menor taxa de erro possível.
Acoplamento Molecular na Pesquisa sobre a COVID-19
Como o tamanho do espaço químico é muito grande, os pesquisadores não conseguem testar todas as moléculas possíveis para descobrir quais são eficazes no combate a uma doença específica. Porém, a partir das informações conhecidas sobre a proteína desejada, é possível usar softwares de dinâmica molecular acelerados por GPU para ter uma noção aproximada do comportamento molecular e simular as proteínas desejadas no nível atômico.
Com softwares como o AutoDock, os pesquisadores podem calcular a interação de energia entre um candidato a medicamento e a proteína desejada. Conhecido como acoplamento molecular, esse processo computacionalmente complexo simula milhões de configurações diferentes para encontrar a organização mais favorável de cada molécula para ligação. Com as mais de 27 mil GPUs NVIDIA do supercomputador Summit do Laboratório Nacional de Oak Ridge, os cientistas da Scripps Research puderam testar um bilhão de candidatos a medicamentos em apenas 12 horas. Mesmo com apenas uma GPU NVIDIA, é possível garantir uma aceleração de 230 vezes em comparação com uma CPU.

O Laboratório Nacional Argonne, localizado em Illinois, está acelerando a pesquisa sobre a COVID-19 com um sistema com a GPU NVIDIA A100baseado na arquitetura de referência DGX SuperPOD. Os pesquisadores do Argonne estão simulando 1,5 milhão de átomos da espícula glicoproteica do vírus para agilizar a pesquisa da vacina e prever a capacidade de acoplamento molecular de milhões de candidatos a medicamentos.
Acelerando a Análise de Imagens Biológicas
O processo de descoberta de medicamentos também exige vários testes físicos, o que é um grande desafio para os pesquisadores farmacêuticos. Um método de teste físico consiste na adição de um composto de medicamento a uma célula infectada no laboratório e na observação de mudanças sutis na célula para saber se a ela voltará a ter a aparência de uma célula saudável.
A empresa de biologia digital , sediada em Salt Lake City, usa AI e GPUs NVIDIA para observar essas mudanças sutis nas imagens das células, analisando terabytes de dados toda semana. A empresa divulgou um conjunto de dados de código aberto sobre a COVID-19, compartilhando dados morfológicos de células humanas com pesquisadores que desenvolvem tratamentos para o vírus.
A AI e a computação acelerada continuam acelerando os pipelines de genômica e descoberta de medicamentos. Por isso, a medicina de precisão, que personaliza o plano de tratamento de cada paciente de acordo com informações sobre seu genoma, será uma realidade cada vez mais próxima.
Nesse caso, os sistemas de recomendação, que podem ser criados com o framework de softwares NVIDIA Merlin poderiam ajudar os profissionais de saúde a personalizar o tratamento de um paciente para garantir os melhores resultados e uma recuperação possivelmente mais rápida.
Leia as publicações do blog e acesse o hub de pesquisas sobre a COVID-19
Inscreva-se no boletim informativo de notícias da NVIDIA sobre a área da saúde aqui.
